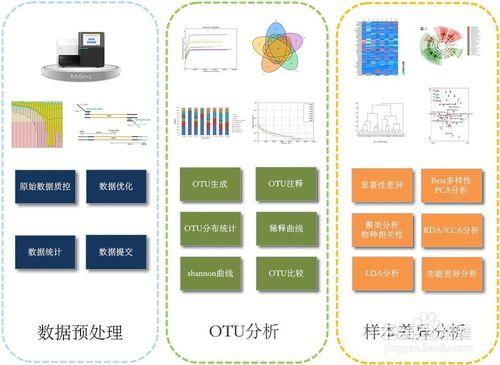
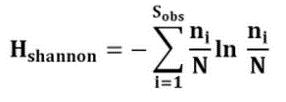微生物群落測序是指對微生物群體進行高通量測序,通過分析測序序列的構成分析特定環境中微生物群體的構成情況或基因的組成以及功能。藉助不同環境下微生物群落的構成差異分析我們可以分析微生物與環境因素或宿主之間的關係,尋找標誌性菌群或特定功能的基因。對微生物群落進行測序包括兩類,一類是通過16s rDNA,18s rDNA,ITS區域進行擴增測序分析微生物的群體構成和多樣性;還有一類是宏基因組測序,是不經過分離培養微生物,而對所有微生物DNA進行測序,從而分析微生物群落構成,基因構成,挖掘有應用價值的基因資源。
以16s rDNA擴增進行測序分析主要用於微生物群落多樣性和構成的分析,目前的生物信息學分析也可以基於16s rDNA的測序對微生物群落的基因構成和代謝途徑進行預測分析,大大拓展了我們對於環境微生物的微生態認知。
目前我們根據16s的測序數據可以將微生物群落分類到種(species)(一般只能對部分菌進行種的鑑定),甚至對亞種級別進行分析,
幾個概念:
16S rDNA(或16S rRNA):16S rRNA 基因是編碼原核生物核糖體小亞基的基因,長度約為1542bp,其分子大小適中,突變率小,是細菌系統分類學研究中最常用和最有用的標誌。16S rRNA基因序列包括9個可變區和10個保守區,保守區序列反映了物種間的親緣關係,而可變區序列則能體現物種間的差異。16S rRNA基因測序以細菌16S rRNA基因測序為主,核心是研究樣品中的物種分類、物種丰度以及系統進化。
OTU:operational taxonomic units (OTUs)在微生物的免培養分析中經常用到,通過提取樣品的總基因組DNA,利用16S rRNA或ITS的通用引物進行PCR擴增,通過測序以後就可以分析樣品中的微生物多樣性,那怎麼區分這些不同的序列呢,這個時候就需要引入operational taxonomic units,一般情況下,如果序列之間,比如不同的 16S rRNA序列的相似性高於97%就可以把它定義為一個OTU,每個OTU對應於一個不同的16S rRNA序列,也就是每個OTU對應於一個不同的細菌(微生物)種。通過OTU分析,就可以知道樣品中的微生物多樣性和不同微生物的丰度。
測序區段:由於16s rDNA較長(1.5kb),我們只能對其中經常變化的區域也就是可變區進行測序。16s rDNA包含有9個可變區,分別是v1-v9。一般我們對v3-v4雙可變區域進行擴增和測序,也有對v1-v3區進行擴增測序。
工具/原料
16s rDNA測序首先需要提取環境樣品的DNA,這些DNA可以來自土壤、糞便、空氣或水體等任何來源。
提取DNA後需要經過質檢和純化,一般16s rDNA測序擴增對DNA的總量要求並不高,總量大於100ng,濃度大於10ng/ul一般都可以滿足要求。如果是來自和寄主共生的環境如昆蟲的腸道微生物,提取時可能包括了寄主本身的大量DNA,對DNA的總量要求會提高。微生物菌群多樣性測序受DNA提取和擴增影響很大,不同的擴增區段和擴增引物甚至PCR循環數的差異都會對結果有所影響。因而建議同一項目不同樣品的都採用相同的條件和測序方法,這樣相互之間才存在可比性。
完成PCR之後的產物一般可以直接上測序儀測序,在上機測序前我們需要對所有樣本進行定量和均一化,通常要進行熒光定量PCR。完成定量的樣品混合後就可以上機測序。
16s rDNA測序目前可以採用多種不同的測序儀進行測序,包括羅氏的454,Illumina的MiSeq,Life的PGM或Pacbio的RSII三代測序儀。不同的儀器各有優缺點,目前最主流的是Illumina公司的MiSeq,因為其在通量、長度和價格三者之間最為平衡。MiSeq測序儀可以產生2x300bp的測序讀長,一次可以產生15Gb的測序數據遠遠大於其他測序儀的測序通量。
方法/步驟
16s rDNA分析基本流程:
原始數據處理:
原始測序數據需要去除接頭序列,並將雙端測序序列進行拼接成單條序列。
根據測序barcode序列區分不同的樣本序列。
過濾低質量序列和無法比對到16s rDNA數據庫的序列。
OTU分類和統計:
OTU(operational taxonomic units) 是在系統發生學研究或群體遺傳學研究中,為了便於進行分析,人為給某一個分類單元(品系,種,屬,分組等)設置的同一標誌。通常按照 97% 的相似性閾值將序列劃分為不同的 OTU,每一個 OTU 通常被視為一個微生物物種。相似性小於97%就可以認為屬於不同的種,相似性小於93%-95%,可以認為屬於不同的屬。樣品中的微生物多樣性和不同微生物的丰度都是基於對OTU的分析。
使用QIIME(version 1.8.0)工具包進行統計註釋。
使用QIIME(version 1.9.0, http://bio.cug.edu.cn/qiime/)的ucluster方法根據97%的序列相似度將所有序列進行同源比對並聚類成operational taxonomic units (OTUs)。然後與數據庫GreenGenes(version gg_13_8, 進行比對,比對方法uclust,identity 0.9 。
然後對每個OTUs進行reads數目統計。
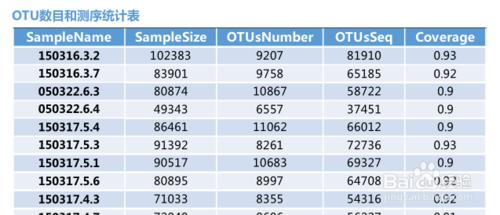
下面的2個表,其中一個表是對每個樣本的測序數量和OTU數目進行統計,並且在表栺中列出了測序覆蓋的完整度(顯示前10個樣本)。
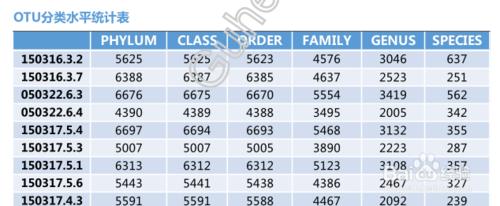
另一個表是對每個樣本在分類字水平上的數量進行統計,並且在表栺中列出了在每個分類字水平上的物種數目(顯示前10個樣本)。
可以看到絕大部分的OTU都分類到了屬(Genus),也有很多分類到了種(Species)。但是仍然有很多無法完全分類到種一級,這是由於環境微生物本身存在非常豐富的多樣性,還有大量的菌仍然沒有被測序和發現。
測序數目統計表主要是對每個樣本的測序數量和OTU數目進行統計,並且在表格中列出了測序覆蓋的完整度(顯示前10個樣本,如果樣本超過10個,請查看結果中otu_stat.txt文件)
其中 SampleName表示樣本名稱;SampleSize表示樣本序列總數;OTUsNumber表示註釋上的OTU數目;OTUsSeq表示註釋上OTU的樣本序列總數。
Coverage是指各樣品文庫的覆蓋率,其數值越高,則樣本中序列沒有被測出的概率越低。該指數實際反映了本次測序結果是否代表樣本的真實情況。
計算公式為:C=1-n1/N 其中n1 = 只含有一條序列的OTU的數目; N = 抽樣中出現的總的序列數目。
分類水平統計表主要是對每個樣本在分類學水平上的數量進行統計,並且在表格中列出了在每個分類學水平上的物種數目(只顯示前10個樣本,如果樣本超過10個,請查看結果中taxon_all.txt文件)
其中SampleName表示樣本名稱;Phylum表示分類到門的OTU數量;Class表示分類到綱的OTU數量;Order表示分類到目的OTU數量;Family表示分類到科的OTU數量;Genus表示分類到屬的OTU數量;Species表示分類到種的OTU數量。
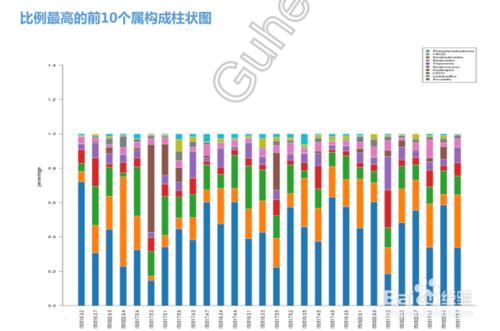
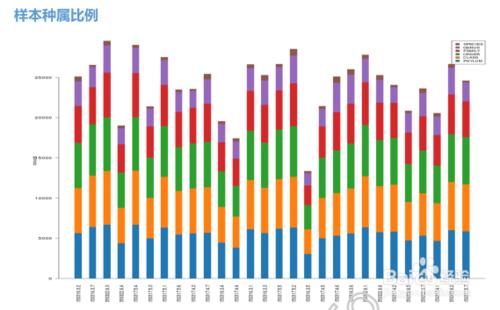
我們還可以對這些種屬的構成進行柱狀圖顯示:
橫座標中每一個條形圖代表一個樣本,縱座標代表該分類層級的序列數目或比例。同一種顏色代表相同的分類級別。圖中的每根柱子中的顏色表示該樣本在不同級別(門、綱、目等)的序列數目,序列數目只計算級別最低的分類,例如在屬中計算過了,則在科中則不重複計算。
Q: 為什麼要選擇V3-V4區的測序長度?為什麼有些文獻是V6區,有什麼區別?
A: 16S rRNA總長約1540 bp,包含 9個可變區。由於高通量測序的測序長度的限制,不可能將16S rRNA的9個可變區全部測序,所以在PCR擴增時往往只能選擇1-3個可變區作為擴增片段。Kozich 等評估了Miseq測序儀分析的不同16S rRNA可變區的準確性發現,測定 V4 區效果最佳。根據我們的測序長度,v3-v4區是最佳選擇。
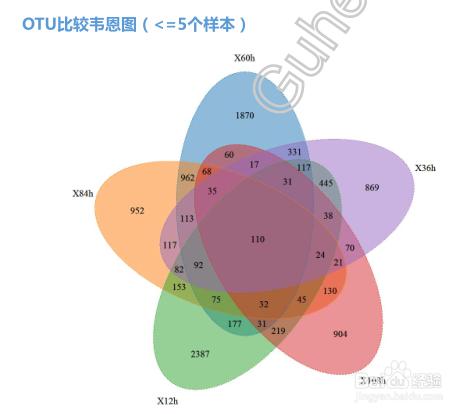
我們還需要對樣本之間或分組之間的OTU進行比較獲得韋恩圖:
注意,韋恩圖目前一般最多隻能顯示5個樣本或分組,過多的樣本無法無法進行韋恩圖繪製
樣品構成丰度:
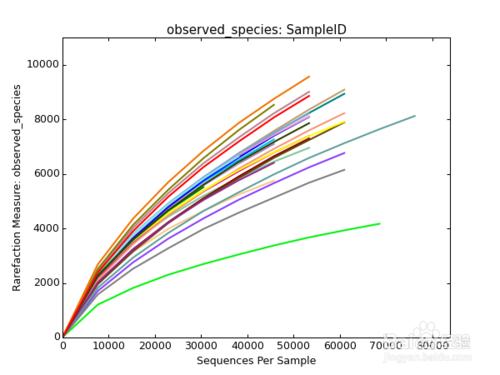
稀釋曲線
微生物多樣性分析中需要驗證測序數據量是否足以反映樣品中的物種多樣性,稀釋曲線(豐富度曲線)可以用來檢驗這一指標。
稀釋曲線是用來評價測序量是否足以覆蓋所有類群,並間接反映樣品中物種的豐富程度。稀釋曲線是利用已測得16S rDNA序列中已知的各種OTU的相對比例,來計算抽取n個(n小於測得reads序列總數)reads時出現OTU數量的期望值,然後根據一組n值(一般為一組小於總序列數的等差數列)與其相對應的OTU數量的期望值做出曲線來。當曲線趨於平緩或者達到平臺期時也就可以認為測序深度已經基本覆蓋到樣品中所有的物種;反之,則表示樣品中物種多樣性較高,還存在較多未被測序檢測到的物種。
下圖中的稀釋曲線
橫座標代表隨機抽取的序列數量;縱座標代表觀測到的OTU數量。樣本曲線的延伸終點的橫座標位置為該樣本的測序數量,如果曲線趨於平坦表明測序已趨於飽和,增加測序數據無法再找到更多的OTU;反之表明不飽和,增加數據量可以發現更多OTU。
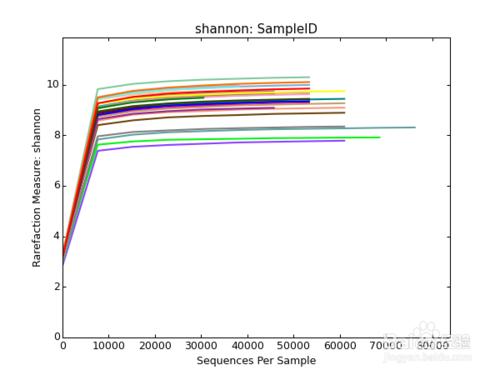
Shannon-Winner曲線
Shannon-Wiener 曲線,是利用shannon指數來進行繪製的,反映樣品中微生物多樣性的指數,利用各樣品的測序量在不同測序深度時的微生物多樣性指數構建曲線,以此反映各樣本在不同測序數量時的微生物多樣性。 當曲線趨向平坦時,說明測序數據量足夠大,可以反映樣品中絕大多數的微生物物種信息。
與上圖一樣,橫座標代表隨機抽取的序列數量;縱座標代表的是反映物種多樣性的Shannon指數。
樣本曲線的延伸終點的橫座標位置為該樣本的測序數量,如果曲線趨於平坦表明測序已趨於飽和,增加測序數據無法再找到更多的OTU;反之表明不飽和,增加數據量可以發現更多OTU。
其中曲線的最高點也就是該樣本的Shannon指數,指數越高表明樣品的物種多樣性越高。
Q: Shannon指數怎麼算的?
A: Shannon指數公式:
其中,Sobs= 實際測量出的OTU數目;ni= 含有i 條序列的OTU數目;N = 所有的序列數。
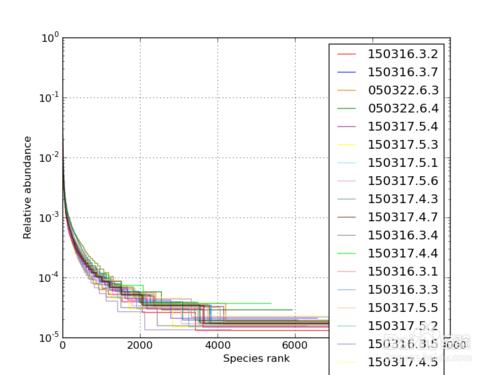
Rank-Abundance曲線
用於同時解釋樣品多樣性的兩個方面,即樣品所含物種的豐富程度和均勻程度。
物種的豐富程度由曲線在橫軸上的長度來反映,曲線越寬,表示物種的組成越豐富;
物種組成的均勻程度由曲線的形狀來反映,曲線越平坦,表示物種組成的均勻程度越高。
一般超過20個樣本圖就會變得非常複雜而且不美觀,所以一般20個樣本以下會做該圖,圖片保存為結果目錄中rank.pdf。
橫座標代表物種排序的數量;縱座標代表觀測到的相對丰度。
樣本曲線的延伸終點的橫座標位置為該樣本的物種數量,如果曲線越平滑下降表明樣本的物種多樣性越高,而曲線快速陡然下降表明樣本中的優勢菌群所佔比例很高,多樣性較低。
Alpha多樣性(樣本內多樣性)
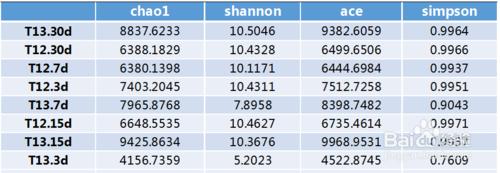
Alpha多樣性是指一個特定區域或者生態系統內的多樣性,常用的度量指標有Chao1 豐富度估計量(Chao1 richness estimator) 、香農 - 威納多樣性指數(Shannon-wiener diversity index)、辛普森多樣性指數(Simpson diversity index)等。
計算菌群丰度:Chao、ace;
計算菌群多樣性:Shannon、Simpson。
Simpson指數值越大,說明群落多樣性越高;Shannon指數越大,說明群落多樣性越高。表中顯示前10個樣本,如果樣本大於10個,詳見結果目錄中的alpha_div.txt。
Q: 能不能解釋下每個指數(如chao1、shannon)?
A: Chao1:是用chao1 算法估計群落中含OTU 數目的指數,chao1 在生態學中常用來估計物種總數,由Chao (1984) 最早提出。Chao1值越大代表物種總數越多。
Schao1=Sobs+n1(n1-1)/2(n2+1)
其中Schao1為估計的OTU數,Sobs為觀測到的OTU數,n1為只有一條序列的OTU數目,n2為只有兩條序列的OTU數目。
Shannon:用來估算樣品中微生物的多樣性指數之一。它與 Simpson 多樣性指數均為常用的反映 alpha 多樣性的指數。Shannon值越大,說明群落多樣性越高。
Ace:用來估計群落中含有OTU 數目的指數,由Chao 提出,是生態學中估計物種總數的常用指數之一,與Chao1 的算法不同。
Simpson:用來估算樣品中微生物的多樣性指數之一,由Edward Hugh Simpson ( 1949) 提出,在生態學中常用來定量的描述一個區域的生物多樣性。Simpson 指數值越大,說明群落多樣性越高。
辛普森多樣性指數=隨機取樣的兩個個體屬於不同種的概率
=1-隨機取樣的兩個個體屬於同種的概率
Beta多樣性分析(樣品間差異分析)
Beta多樣性度量時空尺度上物種組成的變化, 是生物多樣性的重要組成部分, 與許多生態學和進化生物學問題密切相關, 因此在最近10年間成為生物多樣性研究的熱點問題之一。
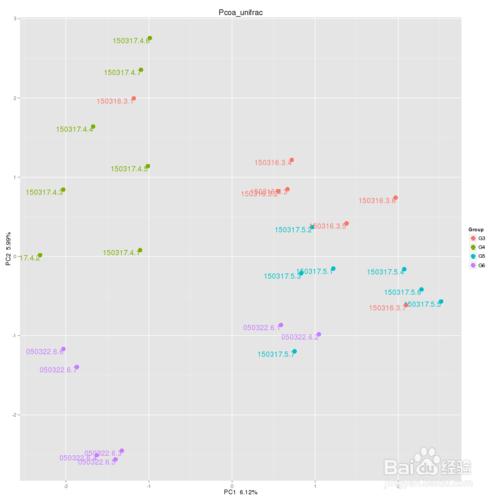
PCoA分析
PCoA(principal co-ordinates analysis)是一種研究數據相似性或差異性的可視化方法,通過一系列的特徵值和特徵向量進行排序後,選擇主要排在前幾位的特徵值,PCoA 可以找到距離矩陣中最主要的座標,結果是數據矩陣的一個旋轉,它沒有改變樣品點之間的相互位置關係,只是改變了座標系統。通過PCoA 可以觀察個體或群體間的差異。
每一個點代表一個樣本,相同顏色的點來自同一個分組,兩點之間距離越近表明兩者的群落構成差異越小。PCoA有多張圖,分別代表的PCoA1-2,2-3,3-1。
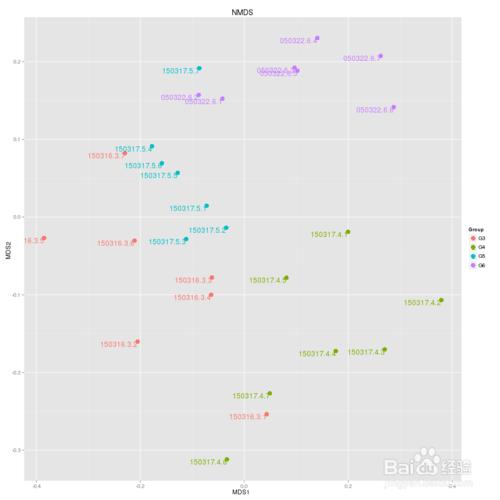
NMDS分析(非度量多維尺度分析)
NMDS(Nonmetric Multidimensional Scaling)常用於比對樣本組之間的差異,可以基於進化關係或數量距離矩陣。
橫軸和縱軸:表示基於進化或者數量距離矩陣的數值 在二維表中成圖。
與PCA分析的主要差異在於考量了進化上的信息。
每一個點代表一個樣本,相同顏色的點來自同一個分組,兩點之間距離越近表明兩者的群落構成差異越小。
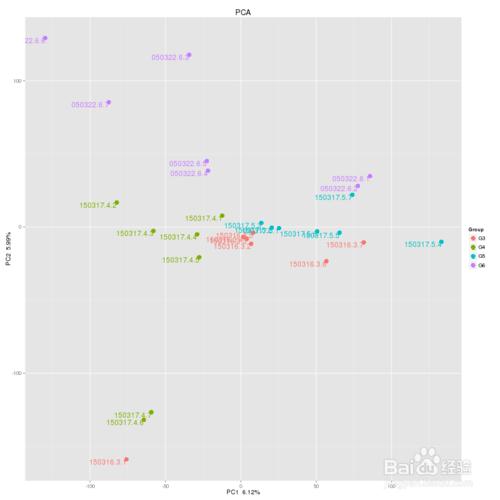
PCA分析
主成分分析PCA(Principal component analysis)是一種研究數據相似性或差異性的可視化方法,通過一系列的特徵值和特徵向量進行排序後,選擇主要的前幾位特徵值,採取降維的思想,PCA 可以找到距離矩陣中最主要的座標,結果是數據矩陣的一個旋轉,它沒有改變樣品點之間的相互位置關係,只是改變了座標系統。詳細關於主成分分析的解釋推薦大家看一篇文章, 。通過PCA 可以觀察個體或群體間的差異。
每一個點代表一個樣本,相同顏色的點來自同一個分組,兩點之間距離越近表明兩者的群落構成差異越小。
以上三個圖可能遇到的問題:
1:PCA,PcoA,NMDS分析分別是基於什麼數據畫的?
回答:PCA,PcoA,NMDS分析均是基於OTU分類taxon數據所畫,用的是R語言Vegan包中的相關函數畫成,其中PcoA與NMDS還要基於樣本之間的距離矩陣才能畫成。
2:PCA分析如果圖中大部分點集中在一起,少數點在很遠的外圍,是什麼原因造成的?
回答:是因為樣本OTU分類時候,少數樣本某些菌含量特別高所造成,導致這些樣本偏離正常範圍,建議單獨拿出這些樣本觀察,看是否是實驗錯誤。
3:PCA分析時,不是有PC1,PC2,PC3三個座標嗎?是給出三張圖嗎?還是三維立體圖?
回答:PCA作圖時,會得出PC1,PC2,PC3三個座標,可以根據PC12,PC13,PC23分別作圖,一般給出的是PC12的圖,當PC12圖質量不好,看不出明顯的樣本分類效果時,可以看PC13或PC23的圖分類是否清晰,也可以用R語言rgl包做出PC123三維圖。
QIIME本身結果中有提供PCA的三維圖結果,可以通過網頁打開。
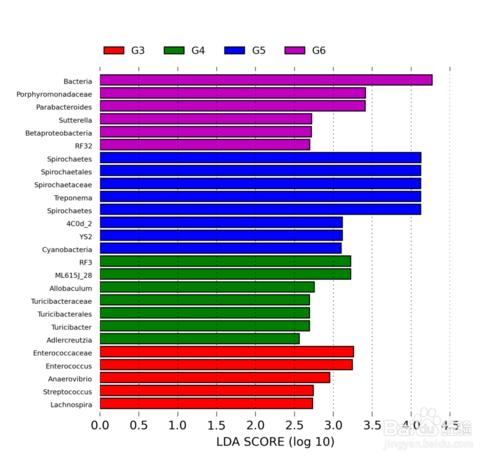
LDA差異貢獻分析
PCA和LDA的差別在於,PCA,它所作的只是將整組數據整體映射到最方便表示這組數據的座標軸上,映射時沒有利用任何數據內部的分類信息,是無監督的,而LDA是由監督的,增加了種屬之間的信息關係後,結合顯著性差異標準測試(克魯斯卡爾-沃利斯檢驗和兩兩Wilcoxon測試)和線性判別分析的方法進行特徵選擇。除了可以檢測重要特徵,他還可以根據效應值進行功能特性排序,這些功能特性可以解釋頂部的大部分生物學差異。詳細說明可以參考這篇文章 cle/details/8071502 。
不同顏色代表不同樣本或組之間的顯著差異物種。使用LefSe軟件分析獲得,其中顯著差異的logarithmic LDA score設為2。
問題:LDA分析有什麼用?
回答:組間差異顯著物種又可以稱作生物標記物(biomarkers),該分析主要是想找到組間在丰度上有顯著差異的物種。
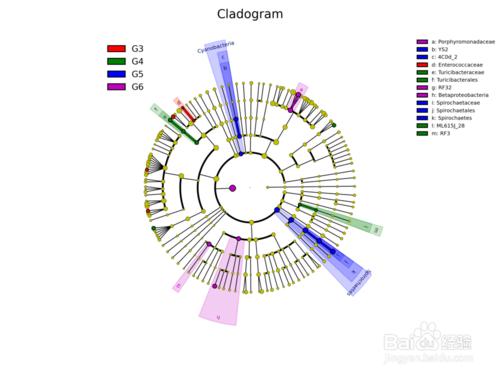
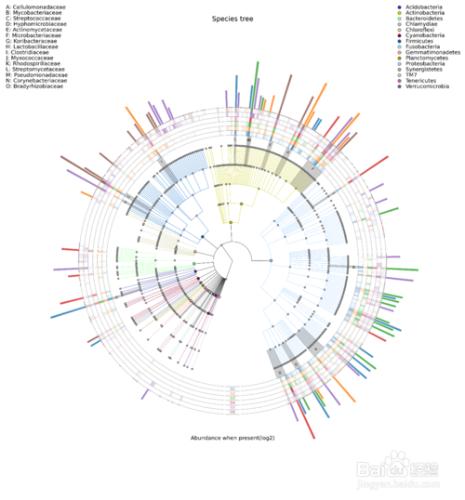
物種進化樹的樣本群落分佈圖
是將不同樣本的群落構成及分佈以物種分類樹的形式在一個環圖中展示。數據經過分析後,將物種分類樹和分類丰度信息通過軟件GraPhlAn( )進行繪製。其目的是將物種之間的進化關係以及不同樣本的物種分佈丰度和最高分佈樣本的信息在一個視覺集中的環圖中一次展示,其提供的信息量較其他圖最為豐富。
中間為物種進化分類樹,不同顏色的分支代表不同的綱(具體的代表顏色見右上角的圖例),之後外圈的灰色標示字母的環表示的是本次研究中比例最高的15個科(字母代表的科參見左上角的圖例)。之後的外圈提供的是熱力圖,如果樣本數<=10個則繪製樣本,如果樣本數超過10個則按照分組繪製,每一環為一個樣本,根據其丰度繪製的熱力圖。最外圈為柱狀圖,繪製的是該屬所佔比例最高的樣本的丰度和樣本顏色(樣本顏色見環最下方的樣本名字的顏色)。其中熱力圖和柱狀圖取值均為原比例值x10000後進行log2轉換後的值
參考文獻:
1. Vazquez-Baeza Y, Pirrung M, Gonzalez A, Knight R. 2013. Emperor: A tool for visualizing high-throughput microbial community data. Gigascience 2(1):16.
2. Legendre, P. and Legendre, L. 1998. Numerical Ecology. Second English Edition. Developments in Environmental Modelling 20. Elsevier, Amsterdam.
3. Segata N, Izard J, Waldron L, et al. Metagenomic biomarker discovery and explanation[J]. Genome Biol, 2011, 12(6): R60.
4. Langille MGI, Zaneveld J, Caporaso JG, McDonald D, Knights D, Reyes JA et al. (2013). Predictive functional profiling of microbial communities using 16S rRNA marker gene sequences. Nat Biotechnol 31: 814–821.
物種相關性分析
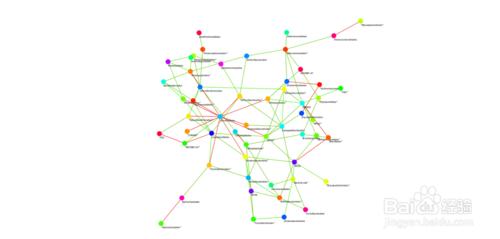
根據各個物種在各個樣品中的丰度以及變化情況,計算物種之間的相關性,包括正相關和負相關。
相關性分析使用CCREPE算法,首先對原始16s測序數據的種屬數量進行標準化,然後進行Spearman和Pearson秩相關分析並進行統計檢驗,計算出各個物種之間的相關性,之後在所有物種中根據simscore絕對值的大小,挑選出相關性最高的前100組數據,基於Cytoscap繪製共表達分析網絡圖,網絡圖採用兩種不同的形式表現出來。
物種相關性網絡圖A:圖中每一個點代表一個物種,存在相關性的物種用連線連接,其中,紅色的連線代表負相關,綠色的先代表正相關,連線顏色的深淺代表相關性的高低。
物種相關性網絡圖B:圖中每一個點代表一個物種,點的大小表示與其他物種的關聯關係的多少,其中與之有相關性的物種數越多,點的半徑和字體越大,連線的粗細代表兩物種之間相關性的大小,連線越粗,相關性越高。
參考文獻:
Schwager E, Weingart G, Bielski C, et al. CCREPE: Compositionality Corrected by Permutation and Renormalization[J]. 2014.
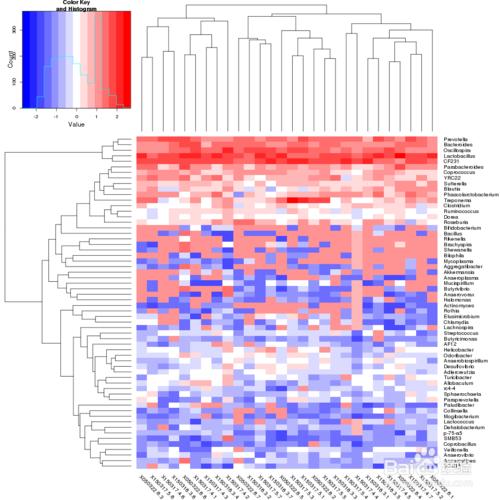
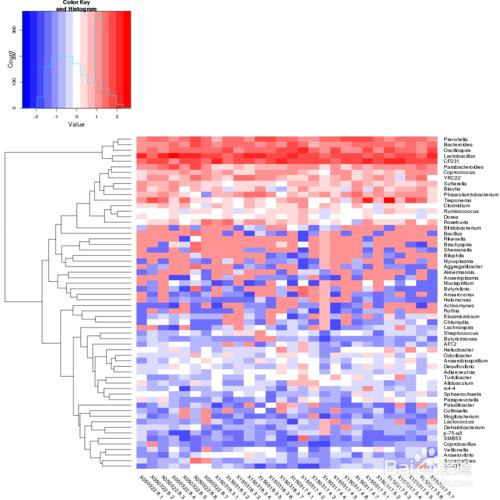
聚類分析
根據OUT數據進行標準化處理(1wlog10)之後,選取數目最多的前60個物種,基於R heatmap進行作圖,熱圖中的每一個色塊代表一個樣品的一個屬的丰度,樣品橫向排列,屬縱向排列,兩個熱圖,差異是是否對樣品進行聚類,從聚類中可以瞭解樣品之間的相似性以及屬水平上的群落構成相似性。
如果聚類結果中出現大面積的白或黑是因為大量的菌含量非常低,導致都沒有數值,可以在繪製之前進行標準化操作,對每一類菌單獨自身進行Z標準化。
群落功能差異分析
通過對已有測序微生物基因組的基因功能的構成進行分析後,我們可以通過16s測序獲得的物種構成推測樣本中的功能基因的構成,從而分析不同樣本和分組之間在功能上的差異(PICRUSt Nature Biotechnology, 1-10. 8 2013)。
通過對宏基因組測序數據功能分析和對應16s預測功能分析結果的比較發現,此方法的準確性在84%-95%,對腸道微生物菌群和土壤菌群的功能分析接近95%,能非常好的反映樣品中的功能基因構成。
為了能夠通過16s測序數據來準確的預測出功能構成,首先需要對原始16s測序數據的種屬數量進行標準化,因為不同的種屬菌包含的16s拷貝數不相同。然後將16s的種屬構成信息通過構建好的已測序基因組的種屬功能基因構成表映射獲得預測的功能結果。(根據屬這個水平,對不同樣本間的物種丰度進行顯著性差異兩兩檢驗,我們這裡的檢驗方法使用STAMP中的two-sample中T-TEST方法,Pvalue值過濾為0.05,作Extent error bar圖。)
此處提供COG,KO基因預測以及KEGG代謝途徑預測。用戶也可自行使用我們提供的文件和軟件(STAMP)對不同層級以及不同分組之間進行統計分析和製圖,以及選擇不同的統計方法和顯著性水平。
參考文獻:
Donovan H. Parks1 , Gene W. Tyson,STAMP: statistical analysis of taxonomic and functional profiles, Bioinformatics (2014) 30(21): 3123-3124.doi:10.1093
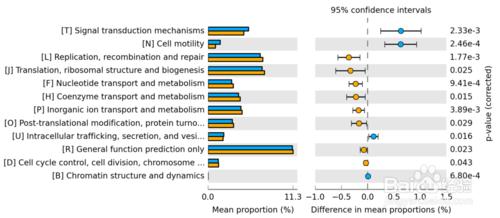
COG構成差異分析圖
圖中不同顏色代表不同的分組,列出了COG構成在組間存在顯著差異的功能分類以及在各組的比例,此外右側還給出了差異的比例和置信區間以及P-value。
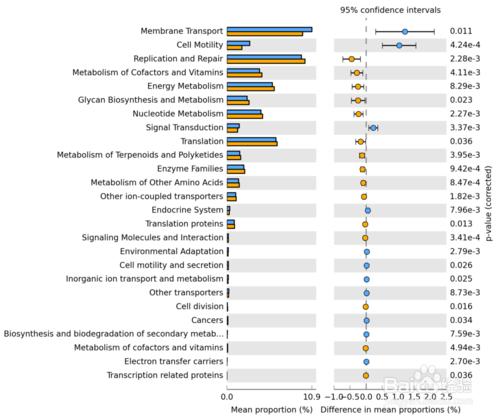
KEGG代謝途徑差異分析圖
通過KEGG代謝途徑的預測差異分析,我們可以瞭解到不同分組的樣品之間在微生物群落的功能基因在代謝途徑上的差異,以及變化的高低。為我們瞭解群落樣本的環境適應變化的代謝過程提供一種簡便快捷的方法。
圖解讀:圖中不同顏色代表不同的分組,列出了在第三層級的構成在組間存在顯著差異的KEGG代謝途徑第三層分類以及在各組的比例,此外右側還給出了差異的比例和置信區間以及P-value。
本例圖所顯示的是第三層級的KEGG代謝途徑的差異分析,也可以針對第二或第一層的分級進行分析。
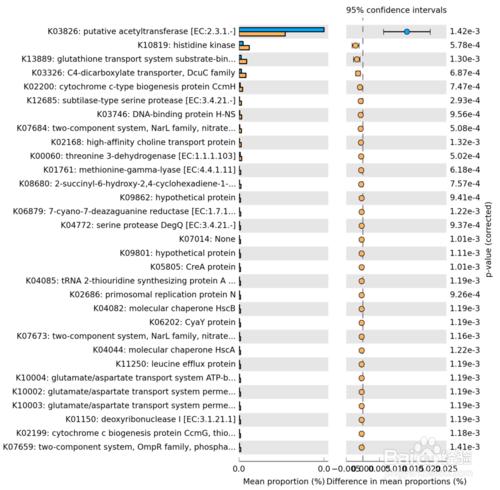
基因的差異分析圖
除了能對大的基因功能分類和代謝途徑進行預測外,我們還能提供精細的功能基因的數量和構成的預測,以及進行樣本間以及組間的差異分析,並給出具有統計意義和置信區間的分析結果。
這一分析將我們對於樣本群落的差異進一步深入到了每一類基因的層面。
圖解讀:圖中不同顏色代表不同的分組,列出了在組間/樣本間存在顯著差異的每一個功能基因(酶)以及在各組的比例,此外右側還給出了差異的比例和置信區間以及P-value。
在獲得標準報告後如果希望單獨修改分組或對某些組之間進行顯著性差異分析,可以使用STAMP軟件在自己的電腦上進行數據分析。STAMP提供了豐富的統計檢驗方法和圖形化結果的輸出。
在使用STAMP之前需要首先準備需要的spf格式文件和樣品分組信息表。在我們的報告中已經將KEGG和KO以及COG的結果文件後經過轉換生成了適用於STAMP軟件打開的spf格式文件,還有對應的分組信息表文件groupfile.txt。
以下是使用STAMP時的一些相關問題,詳細的STAMP使用教程可以參考我們提供的STAMP使用教程。
1、 stamp作圖用的原始數據的來源?
STAMP 可以直接使用來自QIIME的biom文件和PICUST的KEGG和ko 文件,groupfile.txt文件的格式為tab-saperated value (tab鍵隔開的數據)
2、 分組問題:導入數據之後,viewàgroup legend ,在窗口右側會出現分組欄,根據需要進行分組。
3、 Unclassiffied選項中,remain Unclassiffied reads、remove Unclassiffied reads、和use only for calculating frequency profiles 方法的區別?
remain Unclassiffied reads和use only for calculating frequency profiles方法會保留所有的數據,而remove Unclassiffied reads僅僅保留有確定分組信息的數據。
4、 Statistical test 中,Welch’s t-test、t-test、white’s non-parametric t-test的區別,各自優缺點?
為了確保統計學意義和準確度和精確性,需要足夠多的樣本數目,t-test檢驗可以在最少樣本數為4的時候確保高的準確度和精確性。
當兩個樣本之間具有相同方差的時候,用t-test更為準確,當兩個樣本沒有相同方差,Welch’s t-test更為準確。
當樣本數目少於8的時候,可以使用white’s non-parametric t-test,該計算時間較長,當樣本數目過多的時候不宜使用該方法。
5、 Two-group 中type: one side 和 two side 的區別?
One side 只會顯示前一個group與後一個group差異的比例,而two side 兩者之間的比例均會顯示。
6、 STAMP在使用時首先打開了一個分析文件,如果新打開一個可能會導致顯示錯誤?
目前版本的STAMP存在一些小問題,一次分析只能使用一個數據文件,如果要打開新的需要關閉軟件後再打開。
環境因子分析
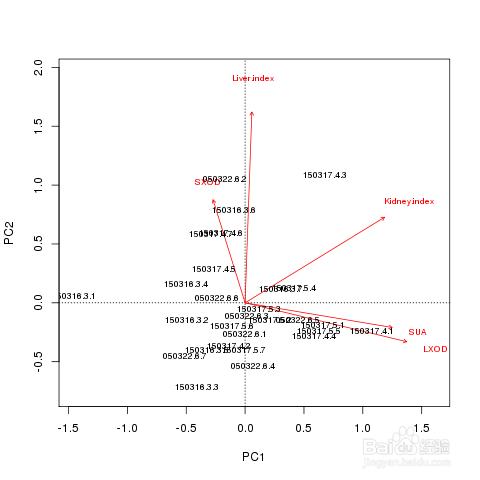
RDA分析
CCA/RDA分析
基於對應分析發展的一種排序方法,將對應分析與多元迴歸分析相結合,每一步計算均與環境因子進行迴歸,又稱多元直接梯度分析。主要用來反映菌群與環境因子之間的關係。
RDA 是基於線性模型,CCA是基於單峰模型。分析可以檢測環境因子、樣品、菌群三者之間的關係或者兩兩之間的關係。
橫軸和縱軸:RDA 和CCA 分析,模型不同,橫縱座標上的刻度為每個樣品或者物種在與環境因子進行迴歸分析計算時產生的值,可以繪製於二維圖形中。
圖解讀:冗餘分析可以基於所有樣品的OTU作圖,也可以基於樣品中優勢物種作圖;
箭頭射線:箭頭分別代表不同的環境因子;
夾角:環境因子之間的夾角為銳角時表示兩個環境因子之間呈正相關關係,鈍角時呈負相關關係。環境因子的射線越長,說明該影響因子的影響程度越大; 不同顏色的點表示不同組別的樣品或者同一組別不同時期的樣品,圖中的拉丁文代表物種名稱,可以將關注的優勢物種也納入圖中; 環境因子數量要少於樣本數量,同時在分析時,需要提供環境因子的數據,比如 pH值,測定的溫度值等。
有其他問題可以聯繫谷禾信息。
注意事項
假設您要對人體腸道微生物菌群進行測序,一般可以選擇對糞便進行取樣。一般建議採取新鮮樣品,因為微生物本身是活的群體,樣品長期保存或不在原環境下保存會改變原有菌群的構成,最終導致我們得到的菌群構成發生偏差。比如糞便樣品如果4度低溫保存了一段時間,則其中部分耐低溫的菌可能仍然在持續繁殖,這樣最終樣品的菌群會發生偏差。如果是送往公司測序也建議首先對樣品進行DNA提取後再寄送,因為原始樣品的寄送過程也可能導致菌群變化。